PySpark DataFrame 을 이용한 탐색적 데이터 분석 및 처리
/* DeepPlay 2022-08-26 */
* 본 포스팅에서는 pyspark 가 설치되어있음을 가정합니다.
최초로 데이터를 받은 이후에, 가장 먼저할 작업은 탐색적 데이터 분석이다. 탐색적 데이터 분석을 통해 각 변수별로 처리 방법을 계획하여, 분석하기 쉬운 형태의 데이터로 만들게 된다. 이 과정에서 변수의 변환, 제거, 생성, 결측값 처리 등의 절차를 수행하게 된다. 본 포스팅은 pyspark 를 통해 데이터를 탐색적으로 확인하는 방법과 간단한 데이터 처리 방법 몇 가지를 다룬다.
pyspark 를 통한 탐색적 분석 문법은 pandas 와 유사한 부분도 있고, 그렇지 않은 부분도 있다. 자주 등장하는 pyspark 의 특이적인 문법이 존재하는데, R, pandas 만 활용해온 사람이라면 이러한 부분에 점점 익숙해질 필요가 있을 것 같다.
필요한 데이터 다운받기: Chronic Kidney Disease Dataset
https://archive.ics.uci.edu/ml/datasets/Chronic_Kidney_Disease
환경세팅
- MAC OS 에서 python 3.8 환경에 pyspark 를 설치하여 실습함
# 데이터 다운로드 받기
# https://www.kaggle.com/datasets/mansoordaku/ckdisease?resource=download
import pyspark
import pandas as pd
from pyspark.sql import SparkSession
from pyspark import SparkContext, SparkConf
import os
import sys
# 환경변수 PYSPARK_PYTHON 와 PYSPARK_DRIVER_PYTHON 의 python path 를 동일하게 맞춰준다.
os.environ['PYSPARK_PYTHON'] = sys.executable
os.environ['PYSPARK_DRIVER_PYTHON'] = sys.executable
# 환경 세팅
conf_spark = SparkConf().set("spark.driver.host", "127.0.0.1") # local 환경임을 명시적으로 지정해준다.
sc = SparkContext(conf=conf_spark)
0. 데이터 로드
pyspark 에서 csv 파일을 load 하는 것은 아래 문법으로 수행할 수 있다.
# data = pd.read_csv("data/kidney_disease.csv") # pandas 에서 csv 파일 읽기
spark = SparkSession.builder.master("local[1]").appName("kidney_disease").getOrCreate()
data = spark.read.option("header", True).csv("data/kidney_disease.csv") # header가 있는 경우, option 구문 추가
data_pd = pd.read_csv("data/kidney_disease.csv")
1. Schema: 데이터 스키마 확인하기
어떤 구조와 변수형으로 데이터가 들어가 있는지 먼저 파악한다.
- 데이터를 직접 추출하여 만들었다면, 각 컬럼이 무엇을 의미하는지 알겠지만..
- 연습을 위한 토이 데이터이거나, 다른 사람한테 받은 데이터라면, 각 컬럼이 무엇을 의미하는지 이해하고 넘어갈 필요가 있다.
- kidney_disease 데이터의 변수명 의미 확인하기: https://archive.ics.uci.edu/ml/datasets/Chronic_Kidney_Disease
# 모든 필드가 string 타입으로 설정되어 있는 것을 확인할 수 있음
data.printSchema()root
|-- id: string (nullable = true)
|-- age: string (nullable = true)
|-- bp: string (nullable = true)
|-- sg: string (nullable = true)
|-- al: string (nullable = true)
|-- su: string (nullable = true)
|-- rbc: string (nullable = true)
2. Show data: 데이터 확인하기
- csv 파일에서 공백인 경우에는 null 값으로 채워졌다는 것을 알 수 있다.
- 각 변수들의 타입을 눈으로 한 번 확인한다.(연속형, 범주형)
data.show()
# data.head() 도 사용 가능함+---+----+-----+-----+----+----+--------+--------+----------+----------+-----+-----+----+-----+----+----+----+-----+----+---+---+---+-----+---+---+--------------+
| id| age| bp| sg| al| su| rbc| pc| pcc| ba| bgr| bu| sc| sod| pot|hemo| pcv| wc| rc|htn| dm|cad|appet| pe|ane|classification|
+---+----+-----+-----+----+----+--------+--------+----------+----------+-----+-----+----+-----+----+----+----+-----+----+---+---+---+-----+---+---+--------------+
| 0|48.0| 80.0| 1.02| 1.0| 0.0| null| normal|notpresent|notpresent|121.0| 36.0| 1.2| null|null|15.4| 44| 7800| 5.2|yes|yes| no| good| no| no| ckd|
| 1| 7.0| 50.0| 1.02| 4.0| 0.0| null| normal|notpresent|notpresent| null| 18.0| 0.8| null|null|11.3| 38| 6000|null| no| no| no| good| no| no| ckd|
3. Size: 데이터의 규모 확인하기
# 400 X 26 size
print(f"Row count: {data.count()}")
print(f"Column count: {len(data.columns)}")Row count: 400
Column count: 264. Univariate distribution (numeric): 단변수 분포 확인하기
#### string -> double 형변환
우선 연속형 변수를 double 형으로 변환해준다. 여기서도 withColumn 구문을 활용할 수 있다.
# 연속형 변수들은 double 로 형변환 수행
numeric_cols = ['age', 'bp', 'sg', 'al', 'su', 'bgr', 'bu', 'sc', 'sod', 'hemo', 'pcv', 'wc', 'rc']
for c in numeric_cols:
data = data.withColumn(c, data[c].cast('double'))#### describe 함수: 기본 통계치 추출
data.describe(numeric_cols).show()+-------+-----------------+-----------------+--------------------+------------------+-------------------+-----------------+------------------+------------------+------------------+------------------+-----------------+------------------+------------------+
|summary| age| bp| sg| al| su| bgr| bu| sc| sod| hemo| pcv| wc| rc|
+-------+-----------------+-----------------+--------------------+------------------+-------------------+-----------------+------------------+------------------+------------------+------------------+-----------------+------------------+------------------+
| count| 391| 388| 353| 354| 351| 356| 381| 383| 313| 348| 329| 294| 269|
| mean|51.48337595907928|76.46907216494846| 1.0174079320113256|1.0169491525423728|0.45014245014245013|148.0365168539326|57.425721784776904|3.0724543080939934|137.52875399361022|12.526436781609195|38.88449848024316| 8406.122448979591| 4.707434944237919|
| stddev|17.16971408926224|13.68363749352527|0.005716616974376756|1.3526789127628445| 1.099191251885407|79.28171423511773|50.503005849222504| 5.741126066859789|10.408752051798777| 2.912586608826765|8.990104814740933|2944.4741904103385|1.0253232655721791|
| min| 2.0| 50.0| 1.005| 0.0| 0.0| 22.0| 1.5| 0.4| 4.5| 3.1| 9.0| 2200.0| 2.1|
| max| 90.0| 180.0| 1.025| 5.0| 5.0| 490.0| 391.0| 76.0| 163.0| 17.8| 54.0| 26400.0| 8.0|
+-------+-----------------+-----------------+--------------------+------------------+-------------------+-----------------+------------------+------------------+------------------+------------------+-----------------+------------------+------------------+
위 결과를 pandas dataframe 으로 변환하고, transpose 를 시켜 보기 쉬운 데이터 형태로 변환한다.
# 첫번째 로우를 header 로 변경
numeric_dist = data.describe(numeric_cols).toPandas().T
new_header = numeric_dist.iloc[0] #grab the first row for the header
numeric_dist = numeric_dist[1:] #take the data less the header row
numeric_dist.columns = new_header #set the header row as the df header
# 인덱스컬럼의 값을 추출해서 새로운 컬럼으로 지정
numeric_dist['col'] = numeric_dist.index.values
numeric_dist.reset_index(drop=True, inplace=True)
#### agg 함수 사용: quantile 값 추출
- 아래와 같이 agg 함수를 활용해 연속형 변수의 quantile 값을 추출할 수 있다.
- pyspark.sql.functions 의 expr 함수를 활용해, agg 함수 내에 원하는 집계 방법을 string 형태로 넣어서 quantile 을 구할 수 있다.
import pyspark.sql.functions as F
perc = pd.DataFrame({'col':[], '%25':[], '%50':[], '%75':[], '%90':[], '%99':[]})
for c in numeric_cols :
result_new = data.agg(
F.expr('percentile(' + c + ',array(0.25))')[0].alias('%25'),
F.expr('percentile(' + c + ',array(0.50))')[0].alias('%50'),
F.expr('percentile(' + c + ',array(0.75))')[0].alias('%75'),
F.expr('percentile(' + c + ',array(0.90))')[0].alias('%90'),
F.expr('percentile(' + c + ',array(0.99))')[0].alias('%99'),
).toPandas()
result_new['col'] = c
perc = pd.concat([perc, result_new], axis=0)
#### 두 결과 테이블 조인하기
- 위 두 테이블을 조인하여 최종적으로 원하는 형태의 연속형 변수 분포 테이블을 만들어 볼 수 있다.
numeric_dist_merge = pd.merge(numeric_dist, perc, how="inner", left_on="col", right_on="col")
numeric_dist_merge[['col', 'count', 'mean', '%25', '%50', '%75', '%90', '%99']]
5. Distribution: 단변수 분포 확인하기 - Categorical variable
#### 범주형 변수에서 unique 한 값 추출하기
- 아래 스크립트에서 rdd.map 이 pyspark 특이적인 문법이라고 볼 수 있다.
ㄴ rdd 는 dataframe 을 rdd 형태로 다룰 것임을 의미하며, rdd 에 lambda 함수를 적용해 결과를 추출한다.
ㄴ collect 함수는 결과를 list 형태로 만들어준다.
ㄴ 이러한 문법은 pandas 나 R 에서는 잘 나오지 않기 때문에 익숙해질 필요가 있을 것 같다.
from pyspark.sql.functions import when
from pyspark.sql import functions as F
# Label 변수 확인하기
print(f"labels: {data.select('classification').distinct().rdd.map(lambda r: r[0]).collect()}")
# data_pd['classification'].unique() # pandas 의 경우는 이와 같이 한다.labels: ['notckd', 'ckd', 'ckd\t']#### 분포 확인하기
- sql.function 내 agg 함수 내에 원하는 집계 (count) 함수를 넣어, 아래와 같이 그룹별 행수를 계산할 수 있다.
result = data.select(['classification']).\
groupBy('classification').\
agg(F.count('classification').alias('user_count')).toPandas()
result
6. Drop useless columns: 컬럼 드랍하기 (Option)
- 필요 없는 컬럼이 있는 경우 drop 함수로 드랍한다.
data = data.drop('pot')
print(f"Row count: {data.count()}")
print(f"Column count: {len(data.columns)}")Row count: 400
Column count: 257. Subsetting: 원하는 행만 남기기
- filter 함수를 통해 원하는 행만 남길 수 있다.
data_ckd = data.filter(data['classification'] == 'ckd')
data_ckd.show()+---+----+-----+-----+----+----+--------+--------+----------+----------+-----+-----+----+-----+----+----+-----+----+---+---+---+-----+---+---+--------------+
| id| age| bp| sg| al| su| rbc| pc| pcc| ba| bgr| bu| sc| sod|hemo| pcv| wc| rc|htn| dm|cad|appet| pe|ane|classification|
+---+----+-----+-----+----+----+--------+--------+----------+----------+-----+-----+----+-----+----+----+-----+----+---+---+---+-----+---+---+--------------+
| 0|48.0| 80.0| 1.02| 1.0| 0.0| null| normal|notpresent|notpresent|121.0| 36.0| 1.2| null|15.4| 44| 7800| 5.2|yes|yes| no| good| no| no| ckd|
| 1| 7.0| 50.0| 1.02| 4.0| 0.0| null| normal|notpresent|notpresent| null| 18.0| 0.8| null|11.3| 38| 6000|null| no| no| no| good| no| no| ckd|
8. Check missing values: 컬럼별 결측값 개수/결측률 확인하기
- 이 데이터에서는 결측값이 null 로 들어가 있다.
- 컬럼별로 결측값 개수를 구하는 것은 다양한 방식으로 구현할 수 있지만, 가장 간단하게 for 을 활용하는 방법은 아래와 같다.
# 각 컬럼별로 결측값 개수를 확인함
# 만약 결측값 코딩이 0으로 되어 있는 경우, filter 조건을 변경하면 된다.
result = pd.DataFrame({'variable':[], 'missing_count':[]})
for col in data.columns:
df_new = pd.DataFrame({'variable':[], 'missing_count':[]})
df_new.at[0, 'variable'] = col
df_new.at[0, 'missing_count'] = data.filter(data[col].isNull()).count()
result = pd.concat([result, df_new], axis=0)
result.astype({'missing_count': 'int32'}) # missing_count 을 int 형으로 변환함
result['missing_rate'] = result['missing_count'] / data.count() # 결측률 계산# 결측률이 높은 순서대로 출력한다.
# rbc 컬럼의 결측율이 38% 로 가장 높은 것을 확인할 수 있다.
result.sort_values(by=['missing_rate'], ascending=False)
9. Replace: 값 치환하기
- 데이터를 다루다보면 종종 값을 치환해야할 경우가 있다. (값이 잘못 들어가 있는 경우, 카테고리를 합치는 경우 등..)
- withColumn 구문과 when 구문을 합쳐여 값 치환을 구현해볼 수 있다.
# label 에 'ckd\t' 로 데이터가 잘못 들어가 있는 것을 확인할 수 있다.
# 이를 when 구문을 통해 원하는 값으로 바꾸어준다.
data = data.withColumn("classification", \
when(data["classification"] == 'ckd\t', 'ckd').otherwise(data["classification"]))
# 재출력하기
print(f"처리후 labels: {data.select('classification').distinct().rdd.map(lambda r: r[0]).collect()}")처리후 labels: ['notckd', 'ckd']
10. Imputation: 평균대치법 (for numeric variable)
- 본 데이터셋은 결측값이 null 로 채워져 있다. 평균 대치법으로 결측값을 처리하자.
- 먼저, 특정 컬럼의 평균을 구하는 함수를 작성한다.
ㄴ pyspark 에서는 이와 관련한 readymade 함수가 없기 때문에 직접 작성한다.
- 여기서도 df.select(avg(df[col]))) 이나 rdd.map 같은 pyspark 특이적 문법이 등장한다.
from pyspark.sql.functions import avg
from pyspark.sql.functions import when, lit
# 컬럼별 평균값 계산하기
def mean_of_pyspark_columns(df, numeric_cols, verbose=False):
col_with_mean=[]
for col in numeric_cols:
mean_value = df.select(avg(df[col]))
avg_col = mean_value.columns[0]
res = mean_value.rdd.map(lambda row : row[avg_col]).collect()
if (verbose==True): print(mean_value.columns[0], "\t", res[0])
col_with_mean.append([col, res[0]])
return col_with_mean각 컬럼들에 대해 평균값을 구한다.
numeric_cols = ['age', 'bp', 'sg', 'al', 'su', 'bgr', 'bu', 'sc', 'sod', 'hemo', 'pcv', 'wc', 'rc']
col_with_mean = mean_of_pyspark_columns(data, numeric_cols)
col_with_mean[['age', 51.48337595907928],
['bp', 76.46907216494846],
['sg', 1.0174079320113256],
['al', 1.0169491525423728],
['su', 0.45014245014245013],
['bgr', 148.0365168539326],
['bu', 57.425721784776904],
['sc', 3.0724543080939934],
['sod', 137.52875399361022],
['hemo', 12.526436781609195],
['pcv', 38.88449848024316],
['wc', 8406.122448979591],
['rc', 4.707434944237919]]각 컬럼별로 반복문을 돌면서, null 인 경우 평균으로 replace 를 수행한다.
# with column when 구문으로 null 일 때, 평균으로 치환한다.
for col, mean in col_with_mean:
data = data.withColumn(col, when(data[col].isNull() == True,
lit(mean)).otherwise(data[col]))
11. Imputation: Mode 대치 (for categorical variable)
- 본 데이터셋은 결측값이 null 로 채워져 있다. 최빈값 대치법으로 결측값을 처리해보자.
- 마찬가지로 컬럼별로 최빈값을 구하는 함수를 작성한다.
def mode_of_pyspark_columns(df, cat_col_list, verbose=False):
col_with_mode=[]
for col in cat_col_list:
#Filter null
df = df.filter(df[col].isNull()==False)
#Find unique_values_with_count
unique_classes = df.select(col).distinct().rdd.map(lambda x: x[0]).collect()
unique_values_with_count=[]
for uc in unique_classes:
unique_values_with_count.append([uc, df.filter(df[col]==uc).count()])
#sort unique values w.r.t their count values
sorted_unique_values_with_count= sorted(unique_values_with_count, key = lambda x: x[1], reverse =True)
if (verbose==True): print(col, sorted_unique_values_with_count, " and mode is ", sorted_unique_values_with_count[0][0])
col_with_mode.append([col, sorted_unique_values_with_count[0][0]])
return col_with_mode각 컬럼들에 대해 최빈값을 구한다.
cat_cols = ['rbc', 'pc', 'pcc', 'ba', 'htn', 'dm', 'cad', 'appet', 'pe', 'ane']
col_with_mode = mode_of_pyspark_columns(data, cat_cols)
col_with_mode[['rbc', 'normal'],
['pc', 'normal'],
['pcc', 'notpresent'],
['ba', 'notpresent'],
['htn', 'no'],
['dm', 'no'],
['cad', 'no'],
['appet', 'good'],
['pe', 'no'],
['ane', 'no']]각 컬럼별로 반복문을 돌면서, null 인 경우 최빈값으로 replace 를 수행한다.
for col, mode in col_with_mode:
data = data.withColumn(col, when(data[col].isNull()==True,
lit(mode)).otherwise(data[col]))결측값 치환이 잘 되었는지를 확인한다. 결측값이 없는 데이터가 되었음을 확인할 수 있다.
# 각 컬럼별로 결측값 개수를 확인함
# 만약 결측값 코딩이 0으로 되어 있는 경우, filter 조건을 변경하면 된다.
result = pd.DataFrame({'variable':[], 'missing_count':[]})
for col in data.columns:
df_new = pd.DataFrame({'variable':[], 'missing_count':[]})
df_new.at[0, 'variable'] = col
df_new.at[0, 'missing_count'] = data.filter(data[col].isNull()).count()
result = pd.concat([result, df_new], axis=0)
result.astype({'missing_count': 'int32'}) # missing_count 을 int 형으로 변환함
result['missing_rate'] = result['missing_count'] / data.count() # 결측률 계산
# 결측률이 높은 순서대로 출력한다.
# rbc 컬럼의 결측율이 38% 로 가장 높은 것을 확인할 수 있다.
result.sort_values(by=['missing_rate'], ascending=False)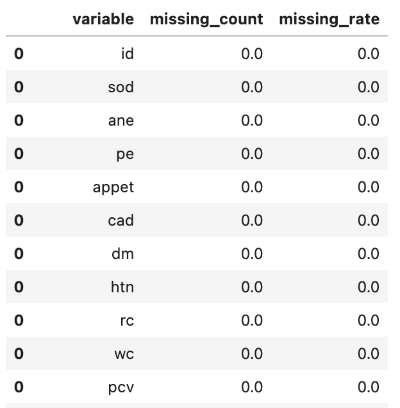
참고 자료
- https://medium.com/@aieeshashafique/exploratory-data-analysis-using-pyspark-dataframe-in-python-bd55c02a2852
- https://towardsdatascience.com/a-brief-introduction-to-pyspark-ff4284701873
A Brief Introduction to PySpark
PySpark is a great language for performing exploratory data analysis at scale, building machine learning pipelines, and creating ETLs for…
towardsdatascience.com
'Tools > Python' 카테고리의 다른 글
| Jupyter notebook에서 외부 파일을 자동으로 갱신하는 방법 (0) | 2022.10.19 |
|---|---|
| Pyspark MLlib 를 활용한 모델링 기초 (w/ Random Forest) (0) | 2022.09.20 |
| PySpark DataFrame 을 사용하는 이유와 pandas 와의 차이점 (0) | 2022.08.26 |
| 아나콘다 python 3 가상환경 세팅하기 (0) | 2022.06.12 |
| Ubuntu 에 Python 새로운 버전 설치하기 (0) | 2019.11.21 |
